Panoramica
I virus sono strumenti eccellenti per le applicazioni di trasferimento genico. La comprensione della biologia molecolare dei virus contribuirà ulteriormente allo sviluppo di strumenti di trasferimento genico ottimizzati. Negli ultimi anni si sono registrati enormi progressi nello sviluppo di tecnologie per l'editing del genoma, in particolare con lo sfruttamento di nucleasi programmabili, come CRISPR/Cas.
Il nostro laboratorio è interessato a sviluppare strumenti molecolari per strategie di terapia genica precise ed efficienti, sfruttando sistemi di distribuzione virali e non virali e nucleasi programmabili.
Direzioni di ricerca
- Sviluppo di strumenti molecolari per un editing del genoma preciso ed efficiente (finanziato da Horizon 20202 EU-FP: UPGRADE e Progetto Sofia):
Questo obiettivo sarà raggiunto attraverso una serie di attività di ricerca:- Sviluppo di CRISPR/Cas inattivi evolutivamente diversificati in strumenti attivi per l'editing di cellule di mammifero: Adatteremo sistemi CRISPR-Cas evolutivamente diversificati per il loro utilizzo in cellule eucariotiche attraverso un processo di mutagenesi casuale e una selezione in vivo. Sono stati riportati diversi sistemi CRISPR-Cas, ma sono inattivi nelle cellule eucariotiche. Queste nuove varianti offrono la possibilità di ampliare la gamma di strumenti per l'editing del genoma in termini di requisiti PAM e dimensioni molecolari.
- Sviluppo di nuove varianti ad alta fedeltà di Cas9 o di ortologhi di Cas9: La piattaforma di screening basata sul lievito sviluppata nel nostro laboratorio (Nature Biotech, 36 265-271 2018) sarà utilizzata per selezionare nuove varianti con specificità migliorata e attività on-target preservata partendo da ortologhi più piccoli.
- Miglioramento della sostituzione genica mediante riparazione omologa diretta (finanziato da Horizon 20202 EU-FP: UPGRADE):
- Identificazione dei fattori cellulari che aumentano l'efficienza dell'editing mirato del genoma. Stiamo eseguendo uno screening cellulare per identificare i fattori che, in combinazione con CRISPR/Cas9 e specifici modelli donatori di DNA, possano migliorare l'efficienza della riparazione diretta per omologia. Vengono utilizzate nuove linee cellulari reporter e tecnologie di screening ad alto contenuto.
- Evoluzione diretta basata sulla tecnologia CRISPR/Cas9
La piattaforma di evoluzione diretta viene impiegata per evolvere nucleasi programmabili. La piattaforma viene anche applicata per invertire l'alterazione funzionale di fattori cellulari mutati nelle malattie genetiche. Questa strategia dovrebbe essere rilevante per quei difetti genetici che non possono essere riparati con strategie di editing del genoma (come delezioni o incompatibilità con i requisiti della PAM).
- Applicazione di strumenti di genome editing per lo sviluppo di strategie di terapia genica (finanziato dalla Fondazione Ricerca Fibrosi Cistica, FFC e dal progetto Sofia):
- Gli strumenti di editing del genoma sono utilizzati nel laboratorio per lo sviluppo di nuove strategie molecolari per il trattamento delle malattie genetiche. Ci occupiamo principalmente di riparare i difetti genetici del gene CFTR nella Fibrosi Cistica sia a livello degli introni che degli esoni del gene mutato. Le strategie di evoluzione diretta di CRISPR/Cas9 vengono applicate anche per i difetti genetici che non possono essere corretti con approcci "standard" di editing del genoma.
- Il laboratorio sta lavorando alla ricerca di strategie di riparazione genomica per il trattamento dei difetti genetici nella sindrome di Cornelia de Lange (CdLS), con particolare attenzione alla rara sostituzione G5483A nel gene NIPBL (progetto Sofia).
Membri del gruppo
- Anna Cereseto, PI
- Antonio Casini, borsista post-dottorato
- Claudia Montagna, borsista post-dottorato
- Gianluca Petris, borsista post-dottorato
- Giulia Maule, dottoranda
- Michele Demozzi, dottorando
Collaborazioni
- Zeger Debyser, KU Leuven, Belgium
- Annarita Miccio, Institute of genetic diseases Imagine, INSERM, Francia
- Thomas Gillingwater, College of Medicine & Veterinary Medicine, University of Edinburgh
- Alessandra Recchia, Università di Modena
Finanziamenti
Bando: PRIN 2022 (D.D. 104/22)
Advancing genome editing technologies for the heart
Anna Cereseto, Responsabile di Unità
Codice Protocollo: 2022Z5PEHM CUP: E53D23005050006

Pubblicazioni selezionate
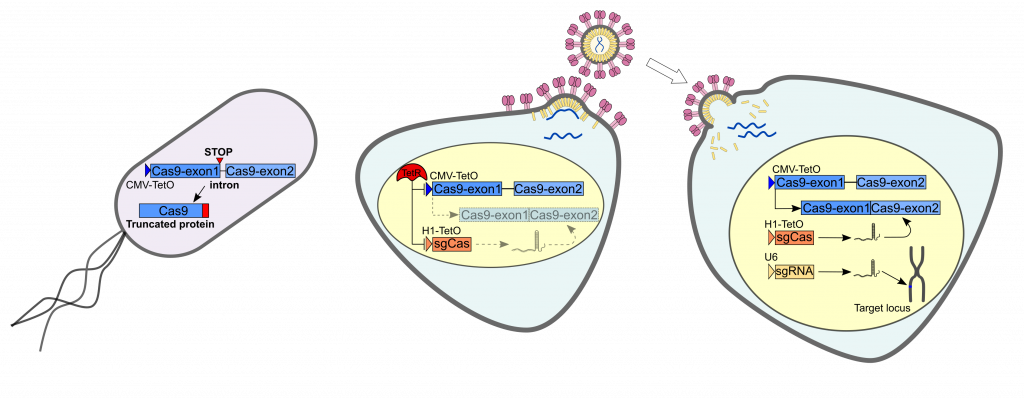
Montagna C, Petris G, Casini A, Maule G, Franceschini GM, Zanella I, Conti L, Arnoldi F, Burrone OR, Zentilin L, Zacchigna S, Giacca M, Cereseto A. VSV-G Enveloped vesicles for traceless delivery of CRISPR-Cas9. Mol. Therapy NA 2018; 12: 453-462.
Casini A, Olivieri M, Petris G, Montagna C, Reginato R, Maule G, Lorenzin F, Prandi D, Romanel A, Demichelis F, Inga A, Cereseto A. In vivo screening of highly specific SpCas9 variants. Nat Biotechnol 2018; 36:265-271.
Romanel A, Garritano S, Stringa B, Blattner M, Dalfovo D, Chakravarty D, Soong D, Cotter KA, Petris G, Dhingra P, Gasperini P, Cereseto A, Elemento O, Sboner A, Khurana E, Inga A, Rubin MA, Demichelis F. Inherited determinants of early recurrent somatic mutations in prostate cancer. Nat Commun 2017;8:48.
Petris G, Casini A, Montagna C, Lorenzin F, Prandi D, Romanel A, Zasso J, Conti L, Demichelis F, Cereseto A. Hit and go CAS9 delivered through a lentiviral based self-limiting circuit. Nat Commun 2017;8:15334.
Cereseto A, Giacca M. Imaging HIV-1 nuclear pre-integration complexes. Methods Mol Biol Clifton NJ 2014;1087:47–54.
Di Primio C, Quercioli V, Allouch A, Gijsbers R, Christ F, Debyser Z, Arosio D, Cereseto A. Single-cell imaging of HIV-1 provirus (SCIP). Proc Natl Acad Sci U S A 2013;110:5636–5641.
Allouch A, Di Primio C, Alpi E, Lusic M, Arosio D, Giacca M, Cereseto A. The TRIM family protein KAP1 inhibits HIV-1 integration. Cell Host Microbe 2011;9:484–495.
Manganaro L, Lusic M, Gutierrez MI, Cereseto A, Del Sal G, Giacca M. Concerted action of cellular JNK and Pin1 restricts HIV-1 genome integration to activated CD4+ T lymphocytes. Nat Med 2010;16:329–333.
Christ F, Thys W, De Rijck J, Gijsbers R, Albanese A, Arosio D, Emiliani S, Rain J-C, Benarous R, Cereseto A, Debyser Z. Transportin-SR2 imports HIV into the nucleus. Curr Biol CB 2008;18:1192–1202.
Albanese A, Arosio D, Terreni M, Cereseto A. HIV-1 pre-integration complexes selectively target decondensed chromatin in the nuclear periphery. PloS One 2008;3:e2413.
