Panoramica
L’epigenetica consente alle cellule di adattarsi alle alterazioni ambientali, causando cambiamenti ereditabili ma reversibili dello stato cellulare. Le perturbazioni del meccanismo epigenetico sono spesso responsabili per le malattie. L’obiettivo del nostro team è quello di studiare come i cambiamenti epigenetici possano avere un impatto sulle cellule staminali, sia in contesti fisiologici sia patologici. Ci proponiamo di analizzare i meccanismi molecolari mediante i quali gli attori della cromatina mediano le risposte epigenetiche ai segnali ambientali nelle cellule staminali. Abbiamo l’intenzione di affrontare questo quesito biologico utilizzando un approccio multidisciplinare il quale, combinando la strategia di tracciamento dinamico di singole cellule con il profiling epigenetico delle cellule, dovrebbe consentire di determinare la rete di modificazioni epigenetiche combinatoriali che determinano uno stato epigenetico simile a quello delle cellule staminali. Sulla stessa linea, siamo anche interessati a comprendere il contributo della riprogrammazione epigenetica indotta dagli oncogeni alla progressione tumorale e alla formazione di metastasi.
Indirizzi di ricerca
- Decifrare il ruolo della riprogrammazione epigenetica nel guidare la plasticità cellulare durante la progressione tumorale e le metastasi
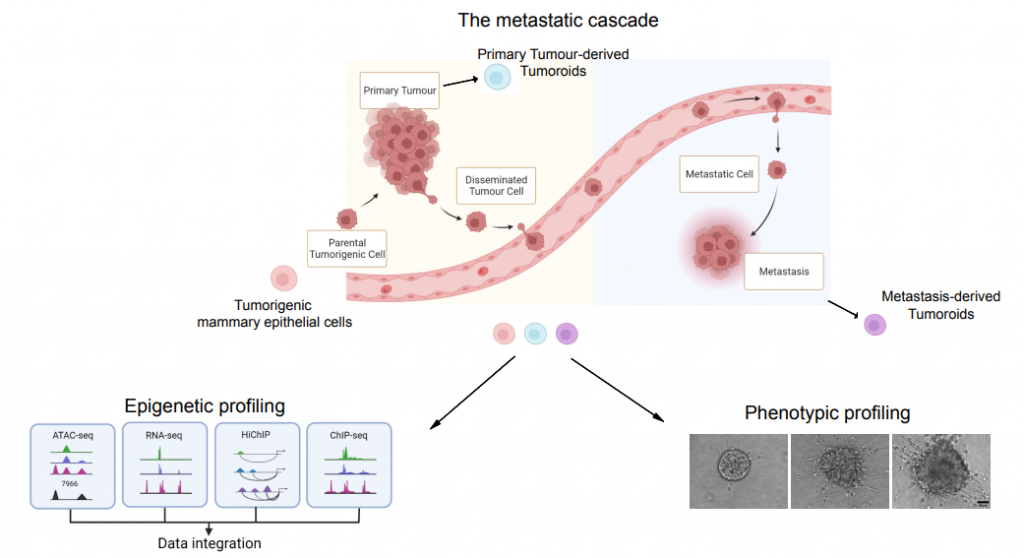
La genomica del cancro ha dimostrato che la progressione del tumore fino alla formazione di metastasi è scarsamente supportata da ulteriori alterazioni genetiche,il che implica che la capacità di adattamento delle cellule tumorali in via di diffusione verso microambienti estranei potrebbe basarsi su alterazioni epigenetiche reversibili. Utilizzando un modello pre-clinico di carcinoma mammario triplo negativo, stiamo studiando il contributo della riprogrammazione epigenetica alla plasticità cellulare guidata dal cancro. Ci chiediamo come lo stato epigenetico oncogenico favorisca l’idoneità delle cellule tumorali durante la progressione tumorale, preservando la memoria degli stimoli precedentemente incontrati attraverso firme molecolari auto-propaganti. Per rispondere a queste domande biologiche, integriamo la profilazione dello stato della cromatina a livello genomico con la topologia del genoma 3D. Stiamo sviluppando approcci computazionali e sperimentali per districare il contributo dei molteplici strati di informazione epigenetica nel garantire la memoria epigenetica nelle cellule tumorali. Come approccio ortogonale, stiamo implementando la metodologia del live imaging per analizzare il ruolo del bookmarking mitotico nel preservare lo stato epigenetico degli enhancer e di altri elementi cis-regolatori.
- Studiare l’interazione tra i condensati della cromatina e la meccanobiologia nelle malattie genetiche rare
La cromatina è organizzata in condensati biomolecolari, i quali compartimentano la funzione del genoma e controllano l'espressione genica. Il nostro obiettivo è risolvere i domini della cromatina mediante l'imaging a super-risoluzione e strumenti basati sull'optogenetica, per determinare il contributo della compartimentazione della cromatina e dell'architettura nucleare all'insorgenza e alla progressione delle malattie genetiche rare. Molte malattie ereditarie derivano da mutazioni nei geni che codificano vari componenti del macchinario epigenetico. Tra le altre, stiamo studiando il ruolo patologico della MLL4 mutata nell'eziologia della sindrome di Kabuki (KS), una malattia con anomalie congenite multiple priva di opzioni terapeutiche. Sviluppando un sistema modello di malattia in vitro, stiamo stabilendo se la patogenesi di questa malattia dipende dalle alterazioni epigenetiche associate a MLL4. Utilizzando tecnologie all'avanguardia, stiamo sviluppando nuovi approcci per definire il ruolo dei condensati di cromatina nello stabilire le proprietà meccaniche nucleari per poi sfruttare nuovi approcci terapeutici per trattare il KS. A questo proposito, siamo interessati a studiare i meccanismi con cui le regioni intrinsecamente disordinate (IDR) di MLL4 guidano l'assemblaggio dei condensati trascrizionali.
Membri del gruppo
- Alessio Zippo, PI
- Sven Beyes, assegnista di ricerca
- Silvia Scalisi, assegnista di ricerca
- Sara Lago, assegnista di ricerca
- Daniela Michelatti, responsabile laboratorio
- Sarah D’Annunzio, dottoranda
- Chiara Bernardis, dottoranda
- Maria Luce Negri, dottoranda
- Leonardo Norelli, dottorando
- Lisa Fol, studentessa universitaria
- Nicole Cona, studente universitario
- Antonio Timpone, studente universitario
Stiamo attualmente cercando candidati altamente motivati per una posizione di dottorando e di assegnista di ricerca interessati a studiare il ruolo della compartimentalizzazione della cromatina nelle malattie.
Collaborazioni
- Prof. Alvaro Rada Iglesias (IBBTEC, Spain)
- Prof. Giorgio Stassi (Università degli Studi di Palermo, Italy)
- Prof. Matilde Todaro (Università degli Studi di Palermo, Italy)
- Mark Neil (Imperial College, UK)
- Prof. Vania Broccoli (San Raffaele Scientific Institute, Italy)
- Prof. Andrea Bassi (Politecnico di Milano, Italy)
- Prof. Ruocco Giancarlo (University of Rome, Italy)
- Prof. Giangaetano Tartaglia (University of Rome, Italy)
- Dr. Paolo Maiuri (IFOM Institute, Italy)
Finanziamenti
Bando: PRIN 2022 (D.D. 104/22)
Dissecting the interplay between chromatin-associated condensates and nuclear mechanical properties in Kabuki Syndrome
Alessio Zippo, Coordinatore
Codice Protocollo: 2022H79275 CUP: E53D23009880001

Pubblicazioni selezionate
MLL4-associated condensates counterbalance Polycomb-mediated nuclear mechanical stress in Kabuki syndrome. Fasciani A, D'Annunzio S, Poli V, Fagnocchi L, Beyes S, Michelatti D, Corazza F, Antonelli L, Gregoretti F, Oliva G, Belli R, Peroni D, Domenici E, Zambrano S, Intartaglia D, Settembre C, Conte I, Testi C, Vergyris P, Ruocco G, Zippo A.
Nat Genet. 2020 Dec;52(12):1397-1411
GADD34 is a modulator of autophagy during starvation. Gambardella G, Staiano L, Moretti MN, De Cegli R, Fagnocchi L, Di Tullio G, Polletti S, Braccia C, Armirotti A, Zippo A, Ballabio A, De Matteis MA, di Bernardo D.
Sci Adv. 2020 Sep 25;6(39):eabb0205.
High-throughput 3D imaging of single cells with light-sheet fluorescence microscopy on chip. Sala F, Castriotta M, Paiè P, Farina A, D'Annunzio S, Zippo A, Osellame R, Bragheri F, Bassi A.
Biomed Opt Express. 2020 Jul 17;11(8):4397-4407.
Truncated BRPF1 cooperates with Smoothened to promote adult Shh medulloblastoma. Aiello G, Ballabio C, Ruggeri R, Fagnocchi L, Morassut I, Caron D, Francesca Garilli, Gianno F, Giangaspero F, Piazza S, Romanel A, Zippo A and Tiberi L.
Cell Rep. 2019 Dec 17;29(12):4036-4052.e10
SETD5 regulates chromatin methylation state and preserves global transcriptional fidelity during brain development and neuronal wiring Sessa A, Fagnocchi L, Mastrototaro G, Massimino L, Zaghi M, Indrigo M, Cattaneo S, Martini D, Gabellini C, Pucci C, Fasciani A, Belli R, Taverna S, Andreazzoli M, Zippo A*, Broccoli V*.
Neuron 2019 Oct 23;104(2):271-289.e13 * Co-senior Authors.
The proneural gene ASCL1 governs the transcriptional subgroup affiliation in glioblastoma stem cells by directly repressing the mesenchymal gene NDRG1. Narayanan A, Gagliardi F, Gallotti AL, Mazzoleni S, Cominelli M, Fagnocchi L, Pala M, Piras IS, Zordan P, Moretta N, Tratta E, Brugnara G, Altabella L, Bozzuto G, Gorombei P, Molinari A, Padua RA, Bulfone A, Politi LS, Falini A, Castellano A, Mortini P, Zippo A, Poliani PL, Galli R.
Cell Death Differ. 2018 Dec 11.
Tumorigenic Cell Reprogramming and Cancer Plasticity: Interplay between Signaling, Microenvironment, and Epigenetics. Vittoria Poli, Luca Fagnocchi and Alessio Zippo.
Stem Cell Int. 2018 May 2;2018:4598195.
Enhancer reprogramming in tumor progression: a new route towards cancer cell plasticity. Fagnocchi L, Poli V, Zippo A.
Cell Mol Life Sci. 2018 Apr 24
MYC-driven epigenetic reprogramming favors the onset of tumorigenesis by inducing a stem cell-like state. Poli V, Fagnocchi L, Fasciani A, Cherubini A, Mazzoleni S, Ferrillo S, Miluzio A, Gaudioso G, Vaira V, Turdo A, Giaggianesi M, Chinnici A, Lipari E, Bicciato S, Bosari S, Todaro M, Zippo A.
Nat Commun. 2018 Mar 9;9(1):1024
A cytosolic Ezh1 isoform modulates a PRC2-Ezh1 epigenetic adaptive response in potmitotic cells. Bodega B, Marasca F, Ranzani V, Cherubini A, Della Valle F, Neguembor MV, Wassef M, Zippo A, Lanzuolo C, Pagani M, Orlando V.
Nat Struct Mol Biol. 2017 May; 24(5):444-452
Fluorescence Resonance Energy Transfer microscopy for measuring chromatin complex structure and dynamics. Alessandro Cherubini and Alessio Zippo.
Methods in Molecular Biology, 2016
Integration of Signalling pathways with the epigenetic machinery in the maintenance of stem cells. Luca Fagnocchi, Mazzoleni Stefania and Alessio Zippo.
Stem Cell International, March 2016.
Lamin A/C sustains PcG proteins architecture maintaining repression of target genes. Cesarini E, Mozzetta C, Marullo F, Gregoretti F, Gargiulo A, Columbaro M, Cortesi A, Antonelli L, Di Pelino S, Squarzoni S, Palacios D, Zippo A, Bodega B, Oliva G, Lanzuolo C.
Journal of Cell Biology 2015 Nov 9;211(3):533-51
FOSL1 controls the assembly of endothelial cells into capillary tubes by direct repression of v and 3 integrin transcription. Evellin S, Galvagni F, Zippo A, Neri F, Orlandini M, Incarnato D, Dettori D, Neubauer S, Kessler H, Wagner EF, Oliviero S.
Mol Cell Biol. 2013 Mar;33(6):1198-209.
MYC regulates the transcription of PRC2 to control the expression of developmental genes in embryonic stem cells. Francesco Neri, Alessio Zippo, Anna Krepelova, Alessandro Cherubini, Marina Rocchigiani, and Salvatore Oliviero.
Mol Cell Biol. 2012 Feb;32(4):840-5
Histone crosstalk between H3S10ph and H4K16ac generates a histone code that mediates transcription elongation. Zippo A, Serafini R, Rocchigiani M, Pennacchini S, Krepelova A, Oliviero S.
Cell. 2009 Sep 18; 138(6):1122-36.
PIM1-dependent phosphorylation of Histone H3 at Serine 10 is required for the transcriptional activation of MYC-target genes. Zippo A, De Robertis A, Serafini R, Oliviero S.
Nat Cell Biol. 2007 Aug; 9(8):932-44.
