Descrizione
 Il nostro laboratorio studia proteine e circuiti di regolazione che promuovono la stabilità cromosomica attraverso le varie fasi della divisione cellulare, utilizzando come modelli di studio il lievito (Saccharomyces cerevisiae) e diverse linee cellulari umane (tumorali e non tumorali). In particolare, ci proponiamo di investigare i processi che avvengono a livello dei centromeri (incluso il complesso del cinetocore che si assembla su essi), del rDNA, e dei telomeri. De-regolazioni di queste attività promuovono l’invecchiamento, l’instabilità genomica e una varietà di malattie incluse il cancro e le sindromi associate all’aneuploidia. Studiare l’attività dei cromosomi durante il ciclo cellulare ci aiuterà a capire meglio l’eziologia di queste malattie ma anche a sviluppare nuovi composti che possono inibirle o revertirle.
Il nostro laboratorio studia proteine e circuiti di regolazione che promuovono la stabilità cromosomica attraverso le varie fasi della divisione cellulare, utilizzando come modelli di studio il lievito (Saccharomyces cerevisiae) e diverse linee cellulari umane (tumorali e non tumorali). In particolare, ci proponiamo di investigare i processi che avvengono a livello dei centromeri (incluso il complesso del cinetocore che si assembla su essi), del rDNA, e dei telomeri. De-regolazioni di queste attività promuovono l’invecchiamento, l’instabilità genomica e una varietà di malattie incluse il cancro e le sindromi associate all’aneuploidia. Studiare l’attività dei cromosomi durante il ciclo cellulare ci aiuterà a capire meglio l’eziologia di queste malattie ma anche a sviluppare nuovi composti che possono inibirle o revertirle.
Per la nostra linea di ricerca utilizziamo tecniche di genetica, biochimica e proteomica, biologia molecolare, (video)microscopia ad alta risoluzione, bioinformatica, biologia dei sistemi, biologia strutturale, modelli computazionali di interazione farmaco-proteina e sviluppo di composti/farmaci chemiosintetici.
Direzioni della ricerca
-
Regolazione della trascrizione centromerica e dell’attività del cinetocore
Dopo la replicazione del DNA, i cinetocori si assemblano gerarchicamente reclutando 60 (lieviti) o 100 proteine (umani) sulle sequenze centromeriche per permettere l’attacco bi-orientato dei cromosomi ai microtubuli del fuso mitotico e assicurare così la fedele trasmissione del materiale genetico in entrambe le cellule figlie. Utilizzando S. cerevisiae e cellule umane, studiamo come la chinasi/ATPasi RioK1 promuova l'attività dei cinetocori attraverso il ciclo cellulare.
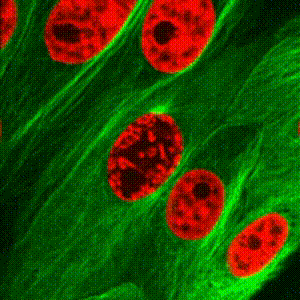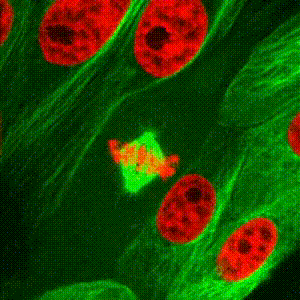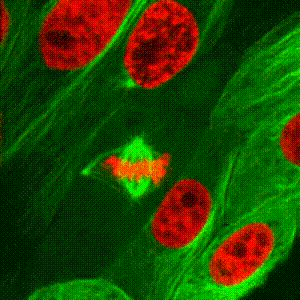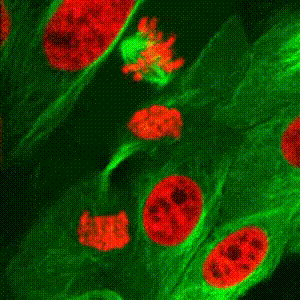
-
Composti anti-tumorali che inibiscono il cinetocore
Molti tumori guidati da una varietà di mutazioni ben note sono suscettibili alla ridotta attività del cinetocore. Utilizzando la struttura del sottocomplesso del cinetocore umano Ndc80, in combinazione con saggi biochimici sviluppati nel nostro laboratorio, abbiamo identificato una serie di piccole molecole che inibiscono l'interazione tra il cinetocore esterno e le estremità dei microtubuli. Attraverso ulteriori ottimizzazioni chemiosintetiche, analisi biologiche e modelli murini di cancro, ci proponiamo di convertire questi composti sia in farmaci antitumorali ad ampio spettro che in strumenti sperimentali che ci consentiranno di indagare attività mitotiche ancora non caratterizzate. -
Studio dell’attività di RioK1
Utilizzando S. cerevisiae e cellule umane studiamo l'attività di RioK1 a livello di DNA, RNA e proteine. Abbiamo dimostrato che in cellule di lievito RioK1 è una chinasi di segnalazione a monte che regola una complessa rete di interazioni comprendente centinaia di geni e proteine. Nello specifico, RioK1 promuove la crescita e la proliferazione in condizioni positive, mentre la inibisce in circostanze negative (stress). Mappando le sue attività durante la progressione del ciclo cellulare sia a livello globale che di singolo gene/RNA/proteina ci proponiamo di descriverne e ampliarne la biologia. Questi studi aiuteranno a comprendere come la sovraespressione di questo enzima, che caratterizza fino al 20% di tutti i tumori maligni, altera la biologia della cellula e innesca l'instabilità cromosomica, culminando nella trasformazione cellulare, nella progressione del cancro e nel processo di metastatizzazione.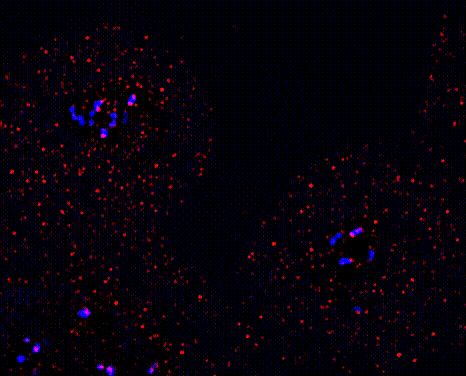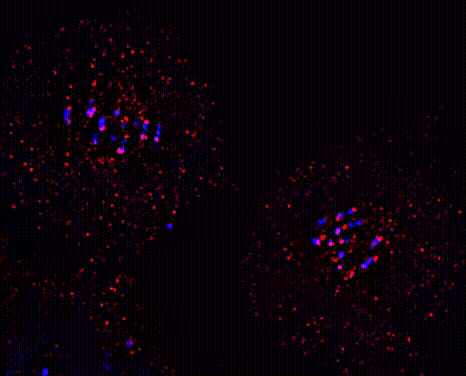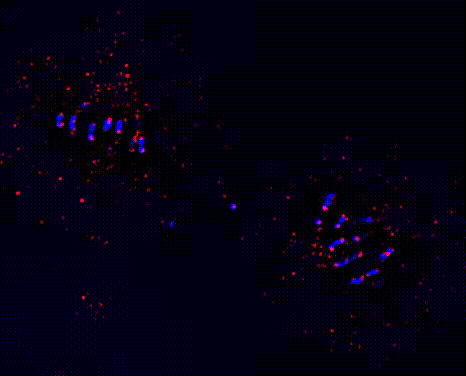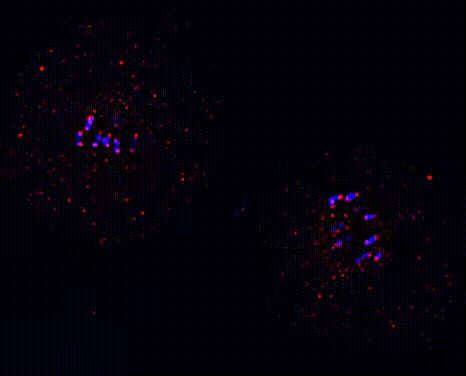
-
Sviluppo di inibitori di RioK1
Utilizzando tecniche computazionali all'avanguardia e un saggio peculiare dell'attività intracellulare di RioK1 umano, stiamo valutando nuovi putativi inibitori di RioK1. Aspiriamo a trasformarli in potenti farmaci per sradicare tumori finora incurabili. Questi piccoli inibitori si dimostreranno anche molto preziosi come nuovi strumenti di ricerca per studiare la biologia di RioK1. -
Studio dell'attività e del rispristino farmacologico della microcefalia guidata da PRUNE1/PPX1
Usando S. cerevisiae come modello genetico studiamo come una mutazione eterozigote dominante-negativa conservata nell'esopolifosfatasi umana PRUNE1 (PPX1 nel lievito) altera la progressione del ciclo cellulare con conseguente sviluppo di microcefalia. Usando il lievito, abbiamo sviluppato un test in tempo reale per identificare i composti che inattivano la mutazione PRUNE1/PPX1, aprendo così una strada verso il futuro trattamento di questa sindrome.
Membri del laboratorio
- Peter De Wulf, PI
- Gloria Allocca, ricercatrice post-dottorale
- Giacomo Biagio De Maio, studente tirocinante magistrale
- Giulia Biasetto, studentessa tirocinante magistrale
- Giorgia Bucciarelli, ricercatrice pre-dottorale
- Michela Damizia, ricercatrice post-dottorale
- Martina Greco, dottoranda
- Joanna Klim, ricercatrice post-dottorale
- Ksenia Smurova, ricercatrice post-dottorale
- Riccardo Tonelli, studente tirocinante triennale
Collaboratori
- Prof. Veronica Ferrucci (Univ. Napoli)
- Prof. Saverio Minucci (IEO Milano)
- Prof. Romano Silvestri (Univ. Roma LaSapienza)
- Prof. Toma Tebaldi (Univ. Trento)
- Prof. Massimo Zollo (Univ. Napoli)
FInanziamenti
- Bando: PRIN 2022 (D.D. 104/22)
Integrating genetic models to mechanistically dissect cytokinesis failure in neurodevelopmental disorders (NDDs)
Peter De Wulf, Responsabile di Unità
Codice Protocollo: 2022T59RWR CUP: E53D23004970006

- Italian Association for Cancer Research (AIRC)
- Fondazione Baggi Sisini
Pubblicazioni selezionate
Damizia M., Moretta G.M., De Wulf P. (2023). The Riok1 network determines p53 activity at multiple levels. Cell Death Discovery, 9(1):410.
Handle F., Puhr M., Gruber M., Ph.D.; Andolfi C.; Schäfer G., Klocker H.; Haybaeck J., De Wulf P., Culig Z. (2023). The oncogenic protein kinase/ATPase RIOK1 is up-regulated via the MYC/E2F transcription factor axis in prostate cancer. American Journal of Pathology, 193(9):1284-97.
Smurova K., Damizia M., Irene C., Stancari S., Berto G., Perticari G., Iacovella M.G., D’Ambrosio I., Giubettini M., Philippe, R., Baggio C., Callegaro E., Casagranda A., Corsini A., Gentile Polese V., Ricci A., Dassi E., De Wulf P. (2023). Rio1 downregulates centromeric RNA levels to promote the timely assembly of structurally fit kinetochores. Nature Communications, 14(1):3172.
Jurková K., De Wulf P., Cusanelli E. (2021). Nuclear periphery and telomere maintenance: TERRA sets the stage. Trends in Genetics, 37:608-611.
Berto G., Ferreira-Cerca S., De Wulf P. (2019). The Rio1 protein kinases/ATPases: conserved regulators of growth, division, and genomic stability. Current Genetics, 65:457-466.
Iacovella M.G., Bremang M., Basha O., Giaco L., Carotenuto W., Golfieri C., Szakal B., Dal MaschioM., Infantino V., Beznoussenko G.V., Chinnu R.J., Visintin C., Mironov A.A., Visintin R., Branzei D., Ferreia-Cerca S., Yeger-Lotem E., De Wulf P. (2018). Integrating Rio1 activities discloses its nutrient-activated network in Saccharomyces cerevisiae. Nucleic Acids Research, 46: 7586-7611.
Smurova K., De Wulf P. (2018). Centromere and pericentromere transcription: roles and regulation... in sickness and in health. Frontiers in Genetics, 9:674.
Iacovella M.G., Golfieri C., Massari L.F., Busnelli S., Pagliuca C., Dal Maschio M., Infantino V., Visintin R., Mechtler K., Ferreira-Cerca S., De Wulf P. (2015). Rio1 promotes rDNA stability and downregulates RNA polymerase I to ensure rDNA segregation. Nature Communications, 6:6643.
Thapa K.S., Oldani A., Pagliuca C., De Wulf P., Hazbun T.R. (2015). The Mps1 kinase modulates the recruitment and activity of Cnn1CENP-T at Saccharomyces cerevisiae kinetochores. Genetics, 200:79-90.
Bock L.J., Pagliuca C., Kobayashi N., Grove R.A., Oku Y., Shrestha K., Alfieri C., Golfieri C., Oldani A., Dal Maschio M., Bermejo R., Hazbun T.R., Tanaka T.U., De Wulf P. (2012). Cnn1 inhibits the interactions between the KMN complexes of the yeast kinetochore. Nature Cell Biology, 14:614-624.
Nguyen T.L., Cera M.T., Pinto A., Lo Presti L., Hamel E., Conti P., Gussio R., De Wulf P. (2012). Evading Pgp activity in drug-resistant cancer cells: a structural and functional study of antitubulin furan metotica compounds. Molecular Cancer Therapeutics, 11:1103-1111.
Screpanti E., Santaguida S., Nguyen T.L., Silvestri R., Gussio R., Musacchio A., Hamel E., De Wulf P. (2010). A screen for kinetochore-microtubule interaction inhibitors identifies novel antitubulin compounds. PLoS One, 5:e11603.
De Wulf P., Montani F., Visintin R. (2009) Protein phosphatases take the mitotic stage. Current Opinion in Cell Biology, 21:806-815.
Pagliuca C., Draviam V.M., Marco E., Sorger P.K., De Wulf P. (2009). Roles for the conserved Spc105p/Kre28p complex in kinetochore-microtubule binding and the spindle assembly checkpoint. PLoS One, 4:e7640.
Fukagawa T., De Wulf P. (2009). Kinetochore composition, formation and organization. In: "The Kinetochore: from Molecular Discoveries to Cancer Therapy". Eds. De Wulf P., Earnshaw W.C. Springer Publ., New York City, p. 133-191.
Ciferri C., Pasqualato S., Screpanti E., Maiolica A., Polka J., DeLuca J.B., De Wulf P., Salek M., Rappsilber J., Moores C.A., Salmon E.D., Musacchio A. (2008). Implications for kinetochore-microtubule attachment from the structure of an engineered Ndc80 complex. Cell, 133:427-439.
Cohen R.L., Espelin C.W., De Wulf P., Sorger P.K., Harrison S.C., Simons K.T. (2008). Structural and functional dissection of Mif2p, a conserved DNA-binding kinetochore protein. Molecular Biology of the Cell, 19:4480-4491.
De Wulf P., Visintin R. (2008). Cdc14B and APC/C tackle DNA damage. Cell, 134:210-212.
Miranda JJ.M., De Wulf P., Sorger P.K., Harrison S.C. (2005). The yeast DASH complex forms closed rings on microtubules. Nature Structural and Molecular Biology, 12:138-143.
De Wulf P., McAinsh A.D., Sorger P.K. (2003). Hierarchical assembly of the budding yeast kinetochore from multiple subcomplexes. Genes and Development, 17:2902-2921.
