Panoramica
La sopravvivenza e la replicazione dei virus dipendono dalla loro capacità di controllare, dirottare e trarre profitto dall'attività della cellula ospite. Decifrare l'interazione tra il virus e la cellula ospite durante il suo ciclo di vita è strumentale non solo per l'identificazione di nuove potenziali opportunità di intervento antivirale, ma anche per una conoscenza meccanicistica più approfondita del meccanismo cellulare. Siamo interessati a entrambi gli aspetti: esplorare il modo in cui i virus interagiscono con i componenti cellulari dell'ospite e migliorare la nostra comprensione dei percorsi dell'ospite coinvolti.
Direzioni di ricerca
La replicazione virale è regolata dall'attività complessa e non sempre ben compresa di proteine virali multifunzionali per ottenere un vantaggio ottimale dal macchinario dell'ospite. D'altra parte, come risultato della sua co-evoluzione con i parassiti virali, la cellula ospite ha sviluppato meccanismi per interferire con la replicazione virale. Stiamo esplorando entrambi gli aspetti di questa relazione ospite-parassita.
-
Le proteine SERINC
Dopo una lunga ricerca per identificare il meccanismo con cui Nef potenzia l'infettività dell'HIV, abbiamo identificato la proteina cellulare SERINC5 come inibitore dei retrovirus (Rosa et al., Nature, 526, 2015). Le proteine SERINC sono proteine transmembrana multipass altamente conservate in tutti gli eucarioti. Abbiamo scoperto che SERINC5 viene incorporato nei virus e interferisce con la capacità delle particelle virali di entrare nelle cellule bersaglio. I nostri studi hanno stabilito che Nef dei lentivirus dei primati (HIV-1, HIV-2 e SIV), così come altri fattori retrovirali dei gammaretrovirus, glicogag (Pizzato M., " PNAS 107, 2010 ), e del virus dell'anemia infettiva equina, S2 (Chande et al., PNAS, 113, 2016); contrastano SERINC5 promuovendo la sua sotto regolazione nel compartimento endosomiale tardivo. L'emergere di un'attività antagonizzante di SERINC in diversi gruppi di retrovirus fornisce un esempio convincente di evoluzione convergente e sottolinea l'importante ruolo antivirale di SERINC5.
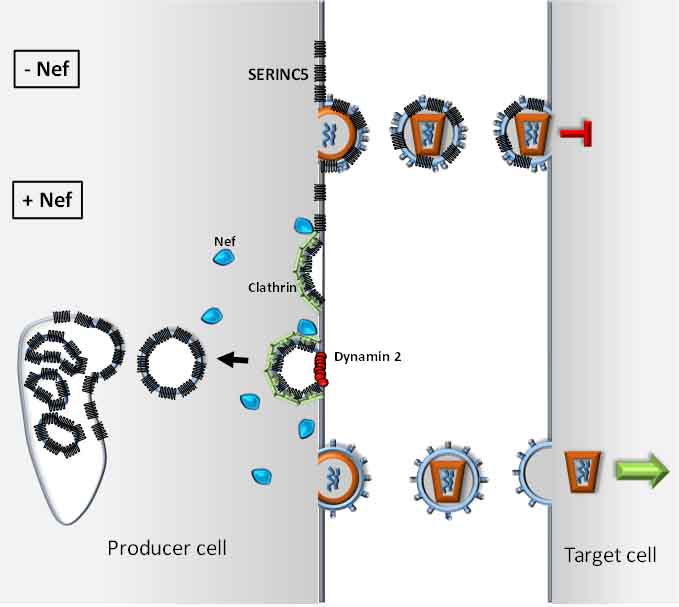
Dopo avere rivelato la struttura tridimesionale delle proteine SERINC (Pye et al., Nat Struct Mol Biol 27, 2020), stiamo investigando non solo la loro attività antivirale, ma anche le loro funzioni nella biologia cellulare e nell’immunità, che rimangono per lo più sconosciute.
Membri del team
- Massimo Pizzato, ricercatore principale
- Francesca Cutrupi, ricercatore post-doc
- Hamza Ur Rehman, dottorando
- Ilary Riedmiller, ricercatore laureato
- Saad Bin Maqsood, ricercatore laureato
Collaborazioni
- Peter Cherepanov, Crick Institute, London
- Jeremy Luban, University of Massachusetts Medical School, USA
- Felipe-Diaz Griffero, Albert Einstein College of Medicine
- Yasuhiro Takeuchi, UCL, London
- Oliver Fackler, University Hospital Heidelberg, Germany
Finanziamenti
Bando: PRIN 2022 (D.D. 104/22)
The cellular function of SERINC proteins and their role in retroviral infection
Massimo Pizzato, Coordinatore
Codice Protocollo: 2022RYNKB5 CUP: E53D23010040006

Pubblicazioni di interesse
Firrito C, Bertelli C, Vanzo T, Chande A, Pizzato M. “SERINC5 as a New Restriction Factor for Human Immunodeficiency Virus and Murine Leukemia Virus.” Annu Rev Virol. 29;5(1):323-340 (2018).
Sood C., Marin M., Chande A., Pizzato M. and Melikyan G.B. “SERINC5 Inhibits HIV-1 Fusion Pore Formation by Promoting Functional Inactivation of Envelope Glycoproteins”. J. Biol. Chem. jbc.M117.777714. (2017).
Chande A, Cuccurullo EC, Rosa A, Ziglio S, Carpenter S, Pizzato M. “S2 from Equine infectious anemia virus is an infectivity factor which counteracts the retroviral inhibitors SERINC5 and SERINC3”. PNAS, 113:13197-13202 (2016).
Trautz B, Pierini V, Wombacher R, Stolp B, Chase AJ, Pizzato M, Fackler OT. “Antagonism of the SERINC5 Particle Infectivity Restriction by HIV-1 Nef Involves Counteraction of Virion-associated Pools of the Restriction Factor.” Journal of virology. JVI.01246-16 (2016)
Heigele A, Kmiec D, Regensburger K, Langer S, Peiffer L, Stürzel CM, Sauter D, Peeters M, Pizzato M, Learn GH, Hahn B, Kirchhoff F. “The Potency of Nef-mediated SERINC5 Antagonism Correlates with the Prevalence of Primate Lentiviruses in the Wild”, Cell Host and Microbe, 20: 381-391 (2016).
Rosa A, Chande A, Ziglio S, De Sanctis V, Bertorelli R, Goh SL, McCauley SM, Nowosielska A, Antonarakis SE, Luban J, Santoni FA, Pizzato M. “HIV-1 Nef promotes infection by excluding SERINC5 from virion incorporation”. Nature, 526: 121-7. (2015).
Pizzato M, McCauley SM, Neagu MR, Pertel T, Firrito C, Ziglio S, Dauphin A, Zufferey M, Berthoux L & Luban J. "Lv4 Is a Capsid-Specific Antiviral Activity in Human Blood Cells That Restricts Viruses of the SIVMAC/SIVSM/HIV-2 Lineage Prior to Integration". PLoS pathogens 11, e1005050 (2015).
Pertel T, Hausmann S, Morger D, Zuger S, Guerra J, Lascano J, Reinhard C, Santoni FA, Uchil PD, Chatel L, Bisiaux A, Albert ML, Strambio-De-Castillia C, Mothes W, Pizzato M, Grutter MG & Luban J. "TRIM5 is an innate immune sensor for the retrovirus capsid lattice". Nature 472, 361-365 (2011).
Lai RP, Yan J, Heeney J, McClure MO, Gottlinger H, Luban J & Pizzato M. "Nef decreases HIV-1 sensitivity to neutralizing antibodies that target the membrane-proximal external region of TMgp41". PLoS pathogens 7, e1002442 (2011).
Pizzato M. "MLV glycosylated-Gag is an infectivity factor that rescues Nef-deficient HIV-1". PNAS 107, 9364-9369 (2010).
Pizzato M, Erlwein O, Bonsall D, Kaye S, Muir D & McClure MO. "A one-step SYBR Green I-based product-enhanced reverse transcriptase assay for the quantitation of retroviruses in cell culture supernatants". Journal of virological methods 156, 1-7 (2009).
Takeuchi Y, McClure MO & Pizzato M. "Identification of gammaretroviruses constitutively released from cell lines used for human immunodeficiency virus research". Journal of virology 82, 12585-12588 (2008).
