Panoramica
Il lavoro nel nostro laboratorio copre un ampio spettro di argomenti relativi alla dinamica delle proteine nell’ambito della fisiologia e delle patologie umane. Indaghiamo questi fenomeni integrando varie tecnologie computazionali, chimiche, biofisiche, biochimiche e cellulari, con l'obiettivo di capitalizzare sulle informazioni ottenute per sviluppare nuove terapie sperimentali per le malattie neurodegenerative.
Ambiti di ricerca
L'espressione e la funzione delle proteine nelle cellule eucariotiche sono processi strettamente armonizzati modulati dalla combinazione di diversi livelli di regolazione, tra cui la trascrizione, l’elaborazione, la stabilità e la traduzione dell'RNA messaggero, nonché l’assemblaggio, la maturazione, la selezione, il riciclo e la degradazione dei polipeptidi. L'integrazione di tutti questi percorsi e del meccanismo di controllo della qualità delle proteine, deputato ad evitare la produzione e l'accumulo di proteine ripiegate in modo aberrante, determina l'omeostasi proteica. Nell'ultimo decennio, lo sviluppo di nuove tecniche informatiche ha aperto la possibilità di studiare i meccanismi biologici a risoluzione atomica con modelli basati sulla fisica. Un esempio significativo è la ricostruzione dei percorsi di ripiegamento delle proteine a risoluzione atomica, che ha consentito la caratterizzazione della cinetica di ripiegamento di proteine globulari biologicamente rilevanti. La combinazione di queste innovative tecnologie computazionali con rigorosi approcci sperimentali rivela l'esistenza di stati metastabili non nativi che appaiono transitoriamente lungo il processo di ripiegamento. Il nostro gruppo è principalmente interessato a testare la possibilità che tali intermedi di ripiegamento proteico possano svolgere ruoli in diversi processi biologici, dalla regolazione post-traduzionale dell'espressione proteica ai meccanismi di misfolding proteico rilevanti per diverse malattie umane. Miriamo a sfruttare le informazioni codificate nei percorsi di ripiegamento delle proteine per progettare una generazione completamente nuova di agenti farmacologici in grado di promuovere la degradazione selettiva dei bersagli proteici.
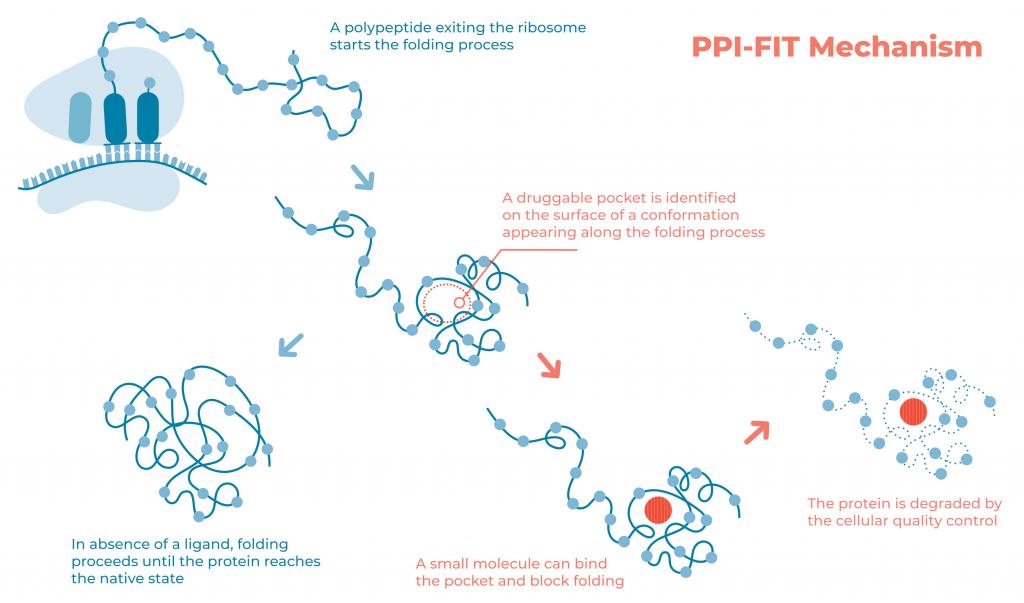
L'immagine illustra la logica alla base del paradigma PPI-FIT (Pharmacological Protein Inactivation by Folding Intermediate Targeting). I polipeptidi che escono dal canale ribosomiale iniziano il processo di ripiegamento. Gli intermedi proteici che compaiono transitoriamente lungo questo processo potrebbero essere presi di mira con piccole molecole che si legano a tasche uniche sulla loro superficie. Un tale evento di legame alla fine interferisce con il processo di ripiegamento delle proteine, spingendo il meccanismo di controllo della qualità delle cellule a reindirizzare il polipeptide verso i percorsi di degradazione. La tecnologia è attualmente sfruttata da Sibylla Biotech (www.sibyllabiotech.it).

Patch della missione spaziale Zeprion2023. L'esperimento mira a ottenere la cristallizzazione di un intermedio di ripiegamento della proteina prionica legata a un suo stabilizzatore farmacologico sulla Stazione Spaziale Internazionale. La missione si basa sull'ipotesi che l'assenza di moti convettivi, ottenuta in condizioni di microgravità, dovrebbe impedire l'aggregazione proteica e favorire la cristallizzazione. L'esperimento è reso possibile dalla collaborazione con l'azienda israeliana SpacePharma (https://www.spacepharma.health/) e dal suo sistema brevettato Lab-On-A-Chip.
COLLABORA CON IL NOSTRO TEAM!
Se sei uno studente al livello di laurea triennale, magistrale o dottorato con un forte interesse per la biologia delle proteine e la scoperta di nuovi farmaci, il nostro gruppo potrebbe essere la soluzione perfetta per te. La dinamica delle proteine è un processo complesso e affrontarlo richiede un'ampia gamma di approcci scientifici. Nel nostro laboratorio avrai l'opportunità di lavorare con un team di ricercatori provenienti da backgrounds scientifici molto diversi, acquisendo esperienza in campi come la biofisica computazionale, la chimica teorica e sintetica, la biochimica e la biologia cellulare. Lavorando con noi, svilupperai una profonda comprensione dei meccanismi molecolari coinvolti nel ripiegamento delle proteine e avrai l'opportunità di utilizzare tecniche avanzate per sondare questo processo e scoprire nuovi farmaci per disturbi neurologici. Unendoti a noi, farai anche parte di una vivace comunità di scienziati appassionati del proprio lavoro. Avrai l'opportunità di partecipare a regolari incontri scientifici, conferenze e seminari, che ti aiuteranno a sviluppare un'ampia rete di collaboratori.
Membri del laboratorio
- Emiliano Biasini, Professore Associato
- Marta Rigoli, Ricercatrice post-doc
- Valerio Bonaldo, Studente di Dottorato
- Ilaria Zeni, Studentessa di Dottorato
- Nicole Innocenti, Studentessa di Dottorato
- Dino Gasparotto, Studente di Dottorato
Collaborazioni
- Ines Mancini, Graziano Lolli, Alberto Inga & Yari Ciribilli, Università di Trento, ITALIA
- Pietro Faccioli, University of Milano-Bicocca, ITALY
- Maria Letizia Barreca, Giuseppe Manfroni & Francesca Fallarino, Università di Perugia, ITALIA
Jesús R. Requena, Università di Santiago de Compostela, SPAGNA
Romolo Nonno, Istituto Superiore di Sanità, ITALIA - Pietro Roversi, CNR, ITALY
- Giovanni Nardo & Valentina Bonetto, Mario Negri Istituto a Milano, ITALY
- Alan Ianeselli, Libera Università di Bolzano, ITALIA
- SpacePharma r&d, ISRAELE
- Sibylla Biotech S.p.A., ITALIA
Pubblicazioni selezionate
Biasini E, Faccioli P. Functional, Pathogenic, and Pharmacological Roles of Protein Folding Intermediates. Proteins. 2023 Feb 13. doi: 10.1002/prot.26479.
https://pubmed.ncbi.nlm.nih.gov/36779817
Spagnolli G, Massignan T, et al. Pharmacological inactivation of the prion protein by targeting a folding intermediate. Commun Biol. 2021 Jan 12;4(1):62. doi: 10.1038/s42003-020-01585-x.
https://pubmed.ncbi.nlm.nih.gov/33437023
Manni G, Lewis V, Senesi M, Spagnolli G, Fallarino F, Collins SJ, Mouillet-Richard S, Biasini E. The cellular prion protein beyond prion diseases. Swiss Med Wkly. 2020 Apr 24;150:w20222. doi: 10.4414/smw.2020.20222.
https://pubmed.ncbi.nlm.nih.gov/32330284
Spagnolli G, Rigoli M, Orioli S, Sevillano AM, Faccioli P, Wille H, Biasini E & Requena JR. Full atomistic model of prion structure and conversion. PLoS Pathog. 2019 Jul 11;15(7):e1007864. doi: 10.1371/journal.ppat.1007864.
https://pubmed.ncbi.nlm.nih.gov/31295325
Rigoli M, Spagnolli G, Faccioli P, Requena JR & Biasini E. Ok Google, how could I design therapeutics against prion diseases? Curr Opin Pharmacol. 2019 May 3;44:39-45. doi: 10.1016/j.coph.2019.03.015.
https://pubmed.ncbi.nlm.nih.gov/31059982
Biasini E. A designer chaperone against prion diseases. Nat Biomed Eng. 2019 Mar;3(3):167-168. doi: 10.1038/s41551-019-0367-6.
https://pubmed.ncbi.nlm.nih.gov/30948815
Per una lista completa: https://www.ncbi.nlm.nih.gov/pubmed/?term=emiliano+biasini
