Panoramica
L'RNA influisce su quasi ogni aspetto dell'espressione genica ed è ormai chiaro che la maggior parte delle malattie genetiche umane sono causate da errori nel metabolismo dell'RNA. È diventato progressivamente evidente che l'RNA non è solo un vettore di informazioni genetiche, ma anche un catalizzatore e una guida per il riconoscimento della sequenza di altre molecole di RNA, responsabile della loro maturazione.
Da alcuni anni, le tecniche basate sull'RNA (RNA interference, RNA antisenso ecc.) stanno diventando sempre più utili nella terapia e nella ricerca applicata. I progressi nella nostra comprensione della biologia dell'RNA hanno permesso di utilizzare l'RNA come strumento per studi funzionali. L’RNA è poi recentemente diventato bersaglio di intervento terapeutico e nuova molecola terapeutica per il trattamento delle malattie umane.
La professoressa Denti ha descritto la sua ricerca sulle terapie a RNA per le malattie neurodegenerative in un'intervista dell'Osservatorio Terapie Avanzate. 
A recent interview with Prof. Michela A. Denti on advantages and pitfalls of RNA-based therapeutics can be found here(in italian).
Una recente intervista con la Prof.ssa Michela A. Denti sui vantaggi e le insidie delle terapie basate sull'RNA può essere trovata qui.
La Professoressa Denti ha parlato Terapie su RNA in una video intervista su RAI 3.
I dettagli della recente partecipazione del laboratorio al consorzio miRNADisEasy, che studia i microRNA come biomarcatori del cancro del polmone, possono essere trovati qui (in sei lingue). Una descrizione della ricerca in corso di laboratorio, il suo significato e le potenziali applicazioni possono essere trovati qui.
 Simone Detassis parla di "Insidie e problemi dell'analisi dell'espressione dei miRNA"
Simone Detassis parla di "Insidie e problemi dell'analisi dell'espressione dei miRNA"
in un webinar per la Oligonucleotide Therapeutics Society il 12 febbraio 2019.
Il laboratorio ha partecipato all'azione COST 17103 "Delivery of Antisense RNA Therapeutics" https://antisenserna.eu.MiRNAQCD: Controllo qualità e diagnosi
MiRNA Un pacchetto R scaricabile per effettuare analisi di controllo qualità, formazione e classificazione su dataset contenenti multipletti MiRNA.
Direzioni di ricerca
- microRNA (miRNA). Sono RNA di circa 21 nucleotidi che controllano lo sviluppo e la differenziazione agendo come regolatori negativi post-trascrizionali dell'espressione di geni bersaglio chiave. Studi recenti hanno dimostrato che vi è un'alterata espressione dei miRNA in diverse neoplasie umane e che i miRNA possono agire come oncogeni o soppressori tumorali. I miRNA sono ora sfruttati anche come biomarcatori diagnostici e prognostici. Il Laboratorio si studia miRNA specificamente sovra- o sotto-espressi nei tumori polmonari, nel melanoma, nei tumori dell'ovaio epiteliale e nelle malattie neurodegenerative (es. demenza frontotemporale) ricerchiando i loro geni bersaglio e studiando il loro ruolo nella patogenesi;
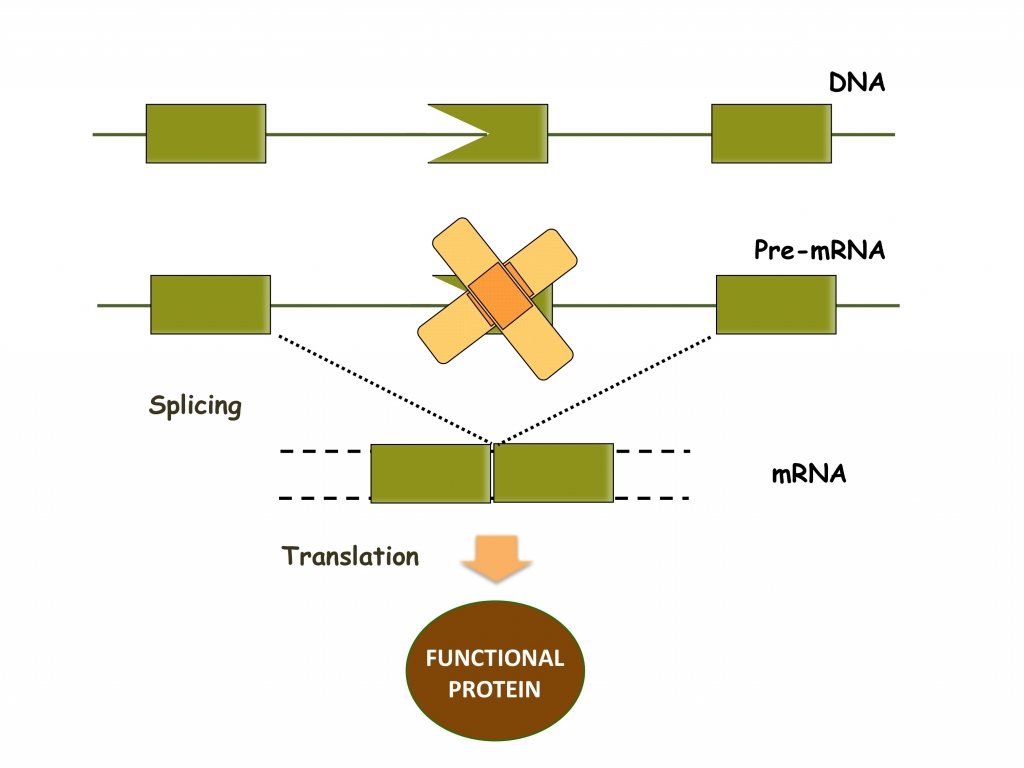
- Correzione terapeutica dello splicing dell’RNA. L'importanza dello splicing alternativo per la diversità del proteoma e il gran numero di malattie genetiche dovute a difetti di splicing richiedono metodi per modulare le decisioni di splicing alternative. Sebbene lo splicing possa essere modulato da oligonucleotidi antisenso, questo approccio si confronta con problemi di veicolazione efficiente e la necessità di somministrazioni ripetute di grandi quantità di oligonucleotidi. Pertanto, abbiamo sviluppato un metodo per indurre il salto efficiente e specifico di un esone con l'aiuto del piccolo RNA nucleare U1 coinvolto nello splicing dell'RNA pre-messaggero nel nucleo. Il salto dell'esone indotto da snRNA U1 antisenso è stato impiegato con successo per elaborare una strategia terapeutica per la distrofia muscolare di Duchenne. Stiamo ora esplorando la possibilità di applicare il salto dell'esone indotto da snRNA U1 antisenso ad altre malattie genetiche, come la demenza frontotemporale, le distrofie retiniche e le malattie metaboliche. Utilizziamo inoltre siRNA isoforma-specifici come possibili terapie basate sulla correzione dello splicing dell’RNA.
Membri del gruppo
- Michela A. Denti, PI
- Ilaria Brentari, candidata a Dottorato di ricerca
- Rafael Filipe Battisti Cavichion, candidato a Dottorato di ricerca
- Elisabetta Callegaro, studentessa MSc
Finanziamenti
- European Commission, Marie Skłodowska-Curie Actions - Research and Innovation Staff Exchange – diaRNAgnosis: A novel platform for the direct profiling of circulating cell-free ribonucleic acids in biofluids.
- Bando: PRIN 2022 (D.D. 104/22)
LuCamiR: Development of a microRNA-based test for the screening of lung cancer
Michela Alessandra Denti, Coordinatrice
Codice Protocollo: 20225AZ22F CUP: E53D23012160006 - Bando: MUR_PNRR_M4C2_Inv 1.3_PE_BaC_MNESYS_Spoke 1 UNIPR
CHARTER: Chemistry and High-frequency oscillatory motion for the formulation in non-viral particles of Antisense RNA Therapeutics for neurodevelopmental disorders
Michela Alessandra Denti, Responsabile scientifica
Codice Progetto: PE00000006 CUP: D93C22000930002 - Bando: PRIN 2022 PNRR (D.D. 1409/22)
MONAD: Combining tailored in silico, in vitro and in vivo models to unveil new NKTR functions associated with rare neurodevelopmental disorders
Michela Alessandra Denti, Responsabile di Unità di Ricerca
Codice Protocollo: P20229PKZC CUP: E53D23015190001

Pubblicazioni selezionate
Articoli
Piscopo P, Margherita G, Manzini V, Zeni A, Castelluzzo M, Fontana F, Talarico G, Castellano AE, Rivabene R, Crestini A, Bruno G, Ricci L, Denti MA. 2023 Identification of miRNAs regulating MAPT expression and their analysis in plasma of patients with dementia. Frontiers in Molecular Neuroscience 16 doi: 10.3389/fnmol.2023.1127163
Alcantara Santos SA, Frediani Portela LM, Lima Camargo AC, Bessi Constantino F, Thassiani Colombelli K, Naia Fioretto M, Mattos R, De Almeida Fantinatti BE, Denti MA, Piazza S, Felisbino SL, Zambrano E, Justulin LA 2022 miR-18a-5p Is Involved in the Developmental Origin of Prostate Cancer in Maternally Malnourished Offspring Rats: A DOHaD Approach. International Journal of Molecular Sciences. 23:14855. doi: 10.3390/ijms232314855.
Covello G, Ibrahim GH, Bacchi N, Casarosa S*, Denti MA* (2022) Exon Skipping Through Chimeric Antisense U1 snRNAs to Correct Retinitis Pigmentosa GTPase-Regulator (RPGR) Splice Defect. Nucleic Acid Therapeutics doi: 10.1089/nat.2021.0053
Mancini M, Grasso M, Muccillo L, Babbio F, Precazzini F, Castiglioni I, Zanetti V, Rizzo F, Pistore C, De Marino MG, Zocchi M, Del Vescovo V, Licursi V, Giurato G, Weisz A, Chiarugi P, Sabatino L, Denti MA*, Bonapace IM* 2021 DNMT3A epigenetically regulates key microRNAs involved in epithelial-to-mesenchymal transition in prostate cancer. Carcinogenesis. 14:1449-60 doi: 10.1093/carcin/bgab101 *corresponding authors
Simone R, Javad F, Emmett W, Wilkins OG, Almeida FL, Barahona-Torres N, Zareba-Paslawska J, Ehteramyan M, Zuccotti P, Modelska A, Siva K, Virdi GS, Mitchell JS, Harley J, Kay VA, Hondhamuni G, Trabzuni D, Ryten M, Wray S, Preza E, Kia DA, Pittman A, Ferrari R, Manzoni C, Lees A, Hardy JA, Denti MA, Quattrone A, Patani R, Svenningsson P, Warner TT, Plagnol V, Ule J, de Silva R. 2021 MIR-NATs repress MAPT translation and aid proteostasis in neurodegeneration. Nature 594:117–23 doi: 10.1038/s41586-021-03556-6
Fantinatti BEA, Perez ES, Zanella BTT, Valente JS, de Paula TG, Mareco EA, Carvalho RF, Piazza S, Denti MA, Dal-Pai-Silva M 2021 Integrative microRNAome analysis of skeletal muscle of Colossoma macropomum (tambaqui), Piaractus mesopotamicus (pacu), and the hybrid tambacu, based on next-generation sequencing data. BMC Genomics 22, 237. doi: 10.1186/s12864-021-07513-5. https://doi.org/10.1186/s12864-021-07513-5
Castelluzzo M, Perinelli A, Detassis S, Denti MA, Ricci L 2020 MiRNA-QC-and-Diagnosis: An R package for diagnosis based on MiRNA expression.
SoftwareX https://doi.org/10.1016/j.softx.2020.100569.
Detassis S, Del Vescovo V, Grasso M, Masella S, Cantaloni C, Cima L, Cavazza A, Graziano P, Rossi G, Barbareschi M, Ricci L, Denti MA. 2020 miR375-3p distinguishes low-grade neuroendocrine from non-neuroendocrine lung tumors in FFPE samples. Front. Mol. Biosci. 7:86. doi: 10.3389/fmolb.2020.00086
Masè M, Grasso M, Avogaro L, Nicolussi Giacomaz N, D’Amato E, Tessarolo F, Graffigna A, Denti MA*, Ravelli F*. 2019 Upregulation of miR-133b and miR-328 in Patients With Atrial Dilatation: Implications for Stretch-Induced Atrial Fibrillation. Front Physiol 10:1133 doi: 10.3389/fphys.2019.01133 *equally contributing
Detassis S, Grasso M, Tabraue-Chávez M, Marin Romero A, Ilyine H, Ress C, Ceriani S, Erspan M, Maglione A, Díaz-Mochón J, Pernagallo S*, Denti MA* 2019 A new platform for the direct profiling of microRNAs in biofluids. Analytical Chemistry. 91:5874-5880 doi: 10.1021/acs.analchem.9b00213 *equally contributing
Grasso M, Piscopo P, Talarico G, Ricci L, Crestini A, Tosto G, Gasparini M, Bruno G, Denti MA*, Confaloni A* 2019 Plasma microRNAs profiling distinguishes patients with frontotemporal dementia from healthy subjects. Neurobiology of Aging 84:240.e1-e12 doi: 10.1016/j.neurobiolaging.2019.01.024 *equally contributing
Piscopo P, Grasso M, Puopolo M, D’Acunto E, Talarico G, Crestini A, Gasparini M, Campopiano R, Gambardella S, Castellano AE, Bruno G, Denti MA*, Confaloni A* 2018 Circulating miR-127-3p as a biomarker for differential diagnosis in Frontotemporal Dementia. J. Alzheimer’s Disease 65: 455-464 doi: 10.3233/JAD-180364 *equally contributing
Pistore C, Giannoni E, Colangelo T, Rizzo F, Magnani E, Muccillo L, Giurato G, Mancini M, Rizzo S, Riccardi M, Sahnane N, Del Vescovo V, Kishore K, Mandruzzato M, Macchi F, Pelizzola M, Denti MA, Furlan D, Weisz A, Colantuoni V, Chiarugi P, Bonapace IM 2017 DNA methylation variations are required for epithelial-to-mesenchimal transition induced by cancer associate fibroblasts in prostate cancer cells Oncogene 36:5551-6 doi: 10.1038/onc.2017.159
Masè M, Grasso M, Avogaro L, D'Amato E, Tessarolo F, Graffigna A, Denti MA*, Ravelli F* 2017 Selection of reference genes is critical for miRNA expression analysis in human cardiac tissue. A focus on atrial fibrillation. Scientific Reports 7:41127 doi: 10.1038/srep41127 *equally contributing
Ferri L, Covello G, Caciotti A, Guerrini R, Denti MA*, Morrone A* 2016 Double-target antisense U1snRNAs correct mis-splicing due to GLA deep intronic mutations in Fabry Disease Molecular Therapy Nucleic Acids 5:e380 doi: 10.1038/mtna.2016.88 *equally contributing
Moncini M, Lunghi M Valmadre A, Grasso M, Del Vescovo V, Riva P, Denti MA, Venturin M 2016 The miR-15/107 family of microRNA genes regulates CDK5R1/p35 with implications for Alzheimer’s disease pathogenesis Mol Neurobiol. doi: 10.1007/s12035-016-0002-4
Piscopo P, Grasso M, Fontana F, Crestini A, Puopolo M, Del Vescovo V, Venerosi A, Calamandrei G, Vencken SF, Greene CM, Confaloni A, Denti MA 2016 Reduced miR-659-3p levels correlate with progranulin increase in hypoxic conditions: implications for frontotemporal dementia Front Mol Neurosci 9:31 doi: 10.3389/fnmol.2016.00031
Ricci L, Del Vescovo V, Cantaloni C, Grasso M, Barbareschi M, Denti MA 2015 Statistical analysis of a Bayesian classifier based on the expression of miRNAs BMC Bioinformatics 16:287 doi: 10.1186/s12859-015-0715-9
Bacchi N, Messina A, Burtscher V, Dassi E, Provenzano G, Bozzi Y, Demontis GC, Koschak A*, Denti MA*, Casarosa S* 2015 A new splicing isoform of Cacna2d4 mimicking the effects of c.2451insC mutation in the retina. Novel molecular and electrophysiological insights. Invest Ophthalmol Vis Sci. 56:4846-56. doi: 10.1167/iovs.15-16410 *equally contributing
Covello G, Siva K, Adami V, Denti MA 2014 An electroporation protocol for efficient DNA transfection in PC12 cells.” Cytotechnology 66:543-553 doi: 10.1007/s10616-013-9608-9
Bisio A, De Sanctis V, Del Vescovo V, Denti MA, Jegga AG, Inga A, Ciribilli Y 2013 Identification of new p53 target microRNAs by bioinformatics and functional analysis” BMC Cancer 13:552 doi: 10.1186/1471-2407-13-552
Del Vescovo V, Meier T, Inga A, Denti MA*, Borlak J* 2013 A Cross-Platform Comparison of Affymetrix and Agilent Microarrays Reveals Discordant miRNA Expression in Lung Tumors of c-Raf Transgenic Mice PLoS ONE 8: e78870. doi: 10.1371/journal.pone.0078870 *equally contributing
Barbareschi M, Cantaloni C, Del Vescovo V, Cavazza A, Monica V, Carella R, Rossi G, Morelli L, Cucino A, Silvestri M, Tirone G, Pelosi G, Graziano P, Papotti M, Dalla Palma P, Doglioni C, Denti MA 2011 Heterogeneity of large cell carcinoma of the lung: an immunophenotypic and miRNA based analysis Am. J. Clin. Pathol. 136:773-782 doi: 10.1309/AJCPYY79XAGRAYCJ
Del Vescovo V, Cantaloni C, Cucino A, Girlando S, Silvestri M, Bragantini E, Fasanella S, Cuorvo LV, Dalla Palma P, Rossi G, Papotti M, Pelosi G, Graziano P, Cavazza A, Denti MA, Barbareschi M 2011 miR-205 expression levels in non-small cell lung cancer do not always distinguish adenocarcinomas from squamous cell carcinomas Am. J. Surg. Pathol. 35:268-275 doi: 10.1097/PAS.0b013e3182068171
Recensioni
Brentari I, Zadorozhna M, Denti MA, Giorgio E 2023 RNA therapeutics for neurological diseases, British Medical Bulletin. ldad010, doi: 10.1093/bmb/ldad010
Brancato V, Brentari I, Coscujuela Tarrero L, Furlan M, Nicassio F*, Denti MA* 2022 News from around the RNA world: new avenues in RNA biology, biotechnology and therapeutics from the 2022 SIBBM meeting. Biology open 11(10) doi:10.1242/bio.059597 *equally contributing
Hammond SM, Aartsma‐Rus A, Alves S, Borgos SE, Buijsen RAM, Collin RWJ, Covello G, Denti MA, Desviat LR, Echevarría L, Foged C, Gaina G, Garanto A, Goyenvalle AT, Guzowska M, Holodnuka I, Jones DR, Krause S, Lehto T, Montolio M, Van Roon‐Mom W, Arechavala‐Gomeza V 2021 Delivery of oligonucleotide‐based therapeutics: challenges and opportunities. EMBO Mol Med 13: e13243. doi:10.15252/emmm.202013243
Precazzini F, Detassis S, Imperatori AS, Denti MA*, Campomenosi P* 2021 Measurements methods for the development of microRNA-based tests for cancer diagnosis Int. J. Mol. Sci. 22:1176 doi:10.3390/ijms22031176 *equally contributing
Detassis S, Grasso M, Del Vescovo V, Denti MA 2017 microRNAs Make the Call in Cancer Personalized Medicine Front. Cell Dev. Biol. 5:86 doi:10.3389/fcell.2017.00086
Godfrey C, Desviat LR, Smedsrod B, Piétri-Rouxel F, Denti MA, Disterer P, Lorain S, Nogales-Gadea G, Sardone V, Anwar R, El Andaloussi S, Lehto T, Khoo B, Brolin Hjortkjaer C, van Roon-Mom WMC, Goyenvalle A, Aartsma-Rus A, Arechavala-Gomeza V 2017 Delivery is key: lessons learnt from developing splice switching antisense therapy EMBO Molecular Medicine doi: 10.15252/emmm.201607199
Fontana F, Siva K, Denti MA 2015 A network in Fronto Temporal Dementia - an RNA perspective Front Mol Neurosci 8:9 doi: 10.3389/fnmol.2015.00009
Grasso M, Piscopo P, Confaloni A, Denti MA 2014 Circulating miRNAs as biomakers for neurodegenerative diseases Molecules 19:6891-6910 doi:10.3390/molecules19056891
Bacchi N, Casarosa S*, Denti MA* 2014 Splicing-correcting therapeutic approaches for retinal dystrophies: where endogenous gene regulation and specificity matter Invest Ophthalmol Visual Sci. 55:3285-94 doi: 10.1167/iovs.14-14544 *equally contributing
Del Vescovo V, Grasso M, Barbareschi M, Denti MA 2014 microRNAs as lung cancer biomarkers World Journal Clinical Oncology 5:604-620 doi: 10.5306/wjco.v5.i4.604
Siva K, Covello G, Denti MA 2014 Exon-skipping Antisense Oligonucleotides to Correct Missplicing in Neurogenetic Diseases” Nucl. Acids Therap. 24: 69-86 doi: 10.1089/nat.2013.0461
Book chapters
Zeni A, Grasso M, Denti MA 2021 Identification of miRNAs Bound to an RNA of Interest by MicroRNA Capture Affinity Technology (miR-CATCH). in “Post-Transcriptional Gene Regulation” Methods in Molecular Biology. Erik Dassi (Ed.) Springer ISBN 978-1-0716-1850-9
Denti MA and Covello G 2017 “Oligonucleotide therapy” in “Safety and Efficacy of Gene-Based Therapeutics for Inherited Disorders” Nicola Brunetti-Pierri (Ed.) Springer ISBN 978-3-319-53455-8 doi: 10.1007/978-3-319-53457-2_9
Del Vescovo V, Denti MA 2015 “microRNA and lung cancer” in “microRNA: Cancer. From molecular biology to clinical practice” Gaetano Santulli (Ed.) Springer ISBN 978-3-319-23729-9
Grasso M, Piscopo P, Crestini A, Confaloni A, Denti MA 2015 “Circulating microRNAs in neurodegenerative diseases” in “Circulating microRNAs in disease diagnostics and their potential biological relevance” Peter Igaz (Ed.) Springer ISBN 978-3-0348-0953-5
Grasso M, Fontana F, Denti MA 2015 “Circulating cell-free microRNAs as biomarkers for neurodegenerative diseases” in “Frontiers in Neurotherapeutics: Mapping Nervous System Diseases via microRNAs” Christian Barbato and Francesca Ruberti (Eds.) CRC Press-Taylor & Francis Group (Boca Raton FL, USA) ISBN 9781482263527

