Panoramica generale
II nostro gruppo si occupa di capire come avviene la divisione cellulare nei batteri di forma sferico-ovoidale (denominati ovococchi) e come questo processo essenziale sia coordinato con l'allungamento laterale, utilizzando come modello lo Streptococcus pneumoniae.
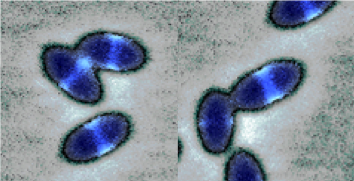
Lo S. pneumoniae (o pneumococco) è un cocco patogeno Gram-positivo, a forma di palla da football americano. Pneumococco vive in intima associazione con l'ospite umano ed è responsabile di più di 1 milione di morti al mondo ogni anno. S. pneumoniae colonizza asintomaticamente il tratto nasofaringeo dei bambini e degli adulti sani, senza effetti negativi apparenti. Tuttavia, S. pneumoniae è anche causa di un ampio spettro di malattie, quali otite media e sinusite, polmonite, batteriemia e meningite. Pneumococco è causa di malattie soprattutto tra gli individui più vulnerabili della società, come i bambini, gli anziani e i soggetti immunocompromessi. La Penicillina e gli altri antibiotici appartenenti alla classe dei beta-lattamici, sono stati usati con successo per anni per la cura delle infezioni da S. pneumoniae. Tuttavia, il numero di ceppi resistenti alla Penicillina sta aumentando drammaticamente a partire dagli anni ’80 e causando seri problemi per quanto riguarda il trattamento delle infezioni sostenute da questo batterio. Inoltre, isolati clinici resistenti anche a altri antibiotici di uso clinico comune continuano ad emergere nonostante l’esistenza di vaccini approvati per la prevenzione delle infezioni da parte di questo batterio. Questo sottolinea la necessità di nuove linee di intervento. Dal 2017, l’Organizzazione Mondiale per la Sanità (WHO) ha infatti incluso lo S. pneumoniae nella lista dei "superbatteri", che rappresentano un problema riguardo l'insorgenza di antibiotico resistenza e per i quali sono urgentemente necessarie ricerca, identificazione e sviluppo di nuovi farmaci antibatterici.
Direzioni di Ricerca
Lo scopo principale della nostra ricerca è quello di elucidare gli aspetti importanti sulla modalità con cui i batteri Gram-positivi di forma sferico-ovoidale coordinano i processi di allungamento e divisione, concentrando la nostra attenzione sulle proteine essenziali coinvolte e sui loro partner di interazione, al fine di determinarne la loro struttura e la funzione.
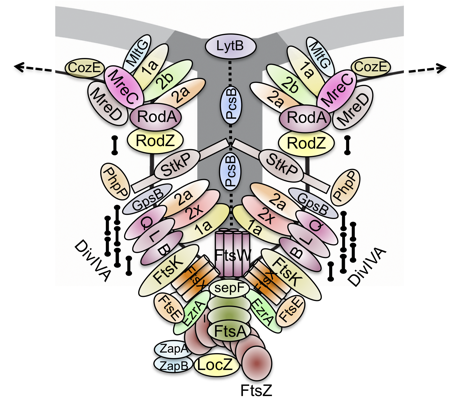
Per ottenere mantenere la loro forma durante il ciclo cellulare, i batteri sfruttano le proteine del loro citoscheletro le quali, tramite polimerizzazione, traducono le interazioni molecolari in una dimensione su più larga scala nella cellula. La maggior parte delle conoscenze che abbiamo riguardo il ciclo cellulare batterico deriva dai più studiati modelli di forma bastoncellare, come Escherichia coli e Bacillus subtilis, i quali crescono principalmente tramite allungamento laterale e successiva divisione, una per ogni generazione, tramite la sintesi di un setto di perpendicolare. In questi modelli di forma bastoncellare, l’allungamento laterale è organizzato dalla proteina MreB, un omologo dell’actina che assembla il complesso biosintetico, noto come elongasoma, responsabile della sintesi laterale della parete cellulare. La divisione cellulare è invece organizzata da FtsZ, un omologo della tubulina che insieme a FtsA, omologo della actina, assembla il complesso biosintetico noto come divisoma responsabile della sintesi della parete cellulare a livello del setto. In modo simile a quanto accade nei bastoncelli, anche S. pneumoniae cresce alternando la sintesi di parete cellulare laterale e a livello del setto, anche se la seconda prevale sulla prima, per ottenere come risultato la caratteristica forma ovale caratteristica di questi batteri, che per questo motivo sono chiamati ovococchi. A sostegno di questo modello, il genoma di S. pneumoniae contieni i geni che codificano le componenti che costituiscono l’elongasoma e il divisoma presenti nei batteri di forma bastoncellare ma, come gli altri cocchi, non possiedono il gene che codifica MreB. Inoltre, anziché avere due complessi biosintetici separati, pneumococco assembla al centro della cellula sia le componenti dell'elongasoma sia quelle del divisoma in un unico complesso, il quale svolge entrambe le funzioni. Questo complesso necessita di un controllo preciso, operato dalla Ser/Thr chinasi di tipo eucariotico, StkP, che integra l’allungamento laterale con la divisione cellulare, con un meccanismo i cui dettagli rimangono ad oggi ancora da chiarire.
La maggior parte del lavoro che stiamo attualmente svolgendo è concentrato sullo studio delle interazioni tra le diverse proteine che compongono gli apparati di crescita e di divisione cellulare di S. pneumoniae, la loro complessa rete di interazioni e le dinamiche alla base della loro localizzazione cellulare. Il nostro scopo è quello di identificare quali siano gli elementi e i motivi chiave presenti in queste proteine, ed eventualmente nella cellula, indispensabili per la vita del batterio.
Questo ci darà anche la possibilità di identificare e validare nuovi bersagli, che potranno essere sfruttati per lo sviluppo di nuovi antibiotici, la cui scoperta è alquanto urgente per contrastare il problema della resistenza.
Membri del gruppo
- Orietta Massidda, PI
- Berenice Furlan, PhD student
- Sarah Costinas, PhD student
- Hamza Ur Rehman, Pre-doc
Collaborazioni
- Malcolm Winkler and Tiffany Tsui, Indiana University, Bloomington, US
- Pavel Branny and Linda Doubravová, Czech Academy of Sciences, Prague, CR
- Waldemar Vollmer, University of Newcastle, Newcastle Upon-Tyne, UK
- William Margolin, McGovern Medical School, Houston, US
- Jan-Willem Veening, University of Lausanne, CH
- Stefano Rossi, Università di Trento
Pubblicazioni selezionate
Lamanna MM, Manzoor I, Joseph M, Ye ZA, Benedet M, Zanardi A, Ren Z, Wang X, Massidda O, Tsui HT, Winkler ME. Roles of RodZ and class A PBP1b in the assembly and regulation of the peripheral peptidoglycan elongasome in ovoid-shaped cells of Streptococcus pneumoniae D39. Mol Microbiol. 2022, 118:336-368. doi: 10.1111/mmi.14969.
Perez AJ, Villicana JB, Tsui HT, Danforth ML, Benedet M, Massidda O, Winkler ME. FtsZ-Ring Regulation and Cell Division Are Mediated by Essential EzrA and Accessory Proteins ZapA and ZapJ in Streptococcus pneumoniae. Front Microbiol, 2021; 12:780864. doi: 10.3389/fmicb.2021.780864.
Vollmer W, Massidda O, Tomasz A. 2019. The Cell Wall of Streptococcus pneumoniae. Microbiol Spectr 7(3). doi: 10.1128/microbiolspec.GPP3-0018-2018.
Cleverley RM, Rutter ZJ, Rismondo J, Corona F, Tsui HT, Alatawi FA, Daniel RA, Halbedel S, Massidda O, Winkler ME, Lewis RJ. 2019. The cell cycle regulator GpsB functions as cytosolic adaptor for multiple cell wall enzymes. Nat Commun, 10:261.
Zheng JJ, Perez AJ, Tsui HT, Massidda O, Winkler ME. 2017. Absence of the KhpA and KhpB (JAG/EloR) RNA-binding proteins suppresses the requirement for PBP2b by overproduction of FtsA in Streptococcus pneumoniae D39. Mol Microbiol, 106:793-814.
Rued BE, Zheng JJ, Mura A, Tsui HT, Boersma MJ, Mazny JL, Corona F, Perez AJ, Fadda D, Doubravová L, Buriánková K, Branny P, Massidda O, Winkler ME. 2017. Suppression and synthetic-lethal genetic relationships of ΔgpsB mutations indicate that GpsB mediates protein phosphorylation and penicillin-binding protein interactions in Streptococcus pneumoniae D39. Mol Microbiol, 103:931-957. Commentary in Lewis RJ. 2017. GpsB the truth is out there. Mol Microbiol, (https://onlinelibrary.wiley.com/doi/full/10.1111/mmi.13612).
Mura A, Fadda D, Perez AJ, Danforth ML, Musu D, Rico AI, Krupka M, Denapaite D, Tsui HT, Winkler ME, Branny P, Vicente M, Margolin W*, Massidda O*. 2016. Roles of the Essential Protein FtsA in Cell Growth and Division in Streptococcus pneumoniae. J Bacteriol, 199:pii:e00608-16. (*co-last corresponding authors).
Holečková N, Doubravová L*, Massidda O*, Molle V, Buriánková K, Benada O, Kofroňová O, Ulrych A, Branny P. 2014. LocZ is a new cell division protein involved in proper septum placement in Streptococcus pneumoniae. MBio, 6:e 01700-14. (*co-corresponding authors).
Massidda, O, Nováková, L, and W Vollmer. 2013. From models to pathogens: How much have we learned about Streptococcus pneumoniae cell division. Env Microbio,15: 3133-57.
Beilharz, K, Nováková, L, Fadda, D, Branny, P, Massidda, O*, Veening, JW*. 2012. Control of cell division in Streptococcus pneumoniae by the conserved Ser/Thr protein kinase StkP. Proc Natl Acad Sci, U.S.A.,109: E905–E913. (*co-last corresponding authors).
Palmieri C, Mingoia, M, Massidda O, Giovanetti E, Varaldo PE. 2012. Streptococcus pneumoniae transposon Tn1545/Tn6003 changes to Tn6002 due to spontaneous excision in circular form of the erm(B)- and aphA3-containing macrolide-aminoglycoside-streptothricin (MAS) element. Antimicrob Agents Chemother, 56:5994–5997.
Maggi, S, Massidda, O, Luzi, G, Fadda, D, Paolozzi, L, Ghelardini, P. 2008. Division protein interaction web: identification of a phylogenetically conserved common interactome between Streptococcus pneumoniae and Escherichia coli. Microbiology (SGM), 154:3042–3052.
Le Gouëllec, A, Roux, L, Fadda, D, Massidda, O, Vernet, T, Zapun, A. 2008. Roles of pneumococcal DivIB in cell division. J Bacteriol, 190:4501–4511.
Fadda, D, Santona, A, D’Ulisse, V, Ghelardini, P, Ennas, MG, Whalen, MB, Massidda, O. 2007. Streptococcus pneumoniae DivIVA: localization and interactions in a MinCD-free context. J Bacteriol, 189:1288–1298. Journal Highlight in "How DivIVA Controls Morphology During Cell Division in S. pneumoniae". Microbe vol 2, 3,2007 (https://iris.unitn.it/retrieve/handle/11572/187531/157696/Fadda%20et%20al_Journal%20Highlight.pdf); Guest Commentary: Vicente M & García-Ovalle M,. 2007. Making a point: the role of DivIVA in "streptococcal polar anatomy”. J Bacteriol, 89:1185-8 (https://jb.asm.org/content/189/4/1185).
Vicente, M, Hodgson, J, Massidda, O, Tonjum, T, Henriques-Normark, B, and Ron, EZ. 2006. The fallacies of hope: will we discover new antibiotics to combat pathogenic bacteria in time? FEMS Microbiol Rev, 30: 841–852.
Smith AM, Feldman, C, Massidda, O, McCarthy, K, Ndiweni, D, Klugman, KP. 2005. Altered PBP 2A and its role in the development of penicillin, cefotaxime, and ceftriaxone resistance in a clinical isolate of Streptococcus pneumoniae. Antimicrob Agents Chemother, 49:2002–2007.
Lara B, Rico AI, Petruzzelli S, Santona A, Dumas J, Biton J, Vicente M, Mingorance J, Massidda O. 2005. Cell division in cocci: localization and properties of the Streptococcus pneumoniae FtsA protein. Mol Microbiol, 55:699–711.
Fadda D, Pischedda C, Caldara F, Whalen MB, Anderluzzi D, Domenici E, Massidda O. 2003. Characterization of divIVA and other genes located in the chromosomal region downstream of the dcw cluster in Streptococcus pneumoniae. J Bacteriol, 185:6209–6214.
Carettoni D, Gómez-Puertas P, Yim, L, Mingorance J, Massidda O, Vicente M, Valencia A, Domenici E, Anderluzzi D. 2003. Phage-display and correlated mutations identify an essential region of subdomain 1C involved in homodimerization of Escherichia coli FtsA. Proteins, 50:192–206.
Massidda O, Anderluzzi D, Friedli L, Feger G 1998. Unconventional organization of the division and cell wall gene cluster of Streptococcus pneumoniae. Microbiology (SGM), 144:3069–3078.
